Les origines de Pubmed
En fait, PubMed n’est pas le début de l’histoire. Son lancement initial en 1996 de même que la mise en ligne de la nouvelle plateforme aujourd’hui, ne sont en effet que des jalons dans la (très) longue histoire de la NLM (National Library of Medicine).
Comme nous n’avons jamais présenté dans BASES la NLM, pourtant un des grands acteurs du secteur, nous rappelons ici les principales étapes de son existence.
La NLM a été créée en 1836 aux Etats-Unis et proposait alors une petite collection d’ouvrages médicaux et de revues. Elle était hébergée dans les locaux de l’United States Army Surgeon General. Elle déménagea plusieurs fois, toujours dans des sites de l’armée, avant de s’installer en 1962 dans le campus du National Institute of Health à Bethesda (MD) où elle se trouve toujours.A partir de 1879, la NLM a publié le bulletin mensuel Index Medicus, un guide des articles publiés dans des milliers de publications.
En 1957, les dirigeants de la NLM commencèrent à penser à une « mécanisation » de l’Index Medicus à la fois pour pouvoir manipuler plus facilement les informations mais aussi pour créer des produits dérivés.
Un cahier des charges fut défini en 1960 suivi d’un appel d’offres remporté par General Electric qui fournit en mars 1963 la banque de données MEDLARS (Medical Literature Analysis and Retrieval System) qui devint réellement opérationnelle en 1964.
A la fin 1971, un maximum de 25 utilisateurs (des bibliothèques spécialisées) pouvaient se connecter simultanément à MEDLINE (MEDLARS ON LINE), nombre augmenté à 900 en janvier 1978 (à comparer aux 3,4 millions d’utilisateurs quotidiens en semaine aujourd’hui !)
Par ailleurs, au début des années 70, les premiers serveurs de banques de données récemment crées à savoir Dialog devenu récemment Dialog Solutions et SDC devenu Orbit racheté par Questel en 1994, se sont intéressés à MEDLINE et ont commencé à l’offrir.
MEDLINE représente l’essentiel de PubMed
MEDLINE est, en fait, un sous-ensemble de PubMed. Selon un article très intéressant Williamson PO, Minter CIJ. Exploring PubMed as a reliable resource for scholarly communications services. J Med Libr Assoc. 2019;107(1):16-29. doi:10.5195/jmla.2019.433 le contenu de MEDLINE représenterait environ 91% du contenu de PubMed, avec une légère tendance à la décroissance mais 82% seulement, d’après un tableau détaillant le contenu de PubMed que nous avons trouvé par ailleurs.Quel que soit le chiffre, cela représente une partie très importante du contenu de PubMed.
Pour limiter ses recherches dans PubMed aux références de Medline, on peut dans sa stratégie de recherche exiger que tous les termes de recherche soient indexés dans le Mesh ou ajouter le suffixe [sb] aux termes de recherche.
On ne trouve dans MEDLINE que des articles indexés avec le MeSH (Medical Subject Heading) un thesaurus sophistiqué des concepts médicaux mis à jour chaque année ce qui entraine une réindexation de l’ensemble de MEDLINE. Ce thesaurus a d’ailleurs été traduit en français par l’INSERM (//mesh.inserm.fr/FrenchMesh). On notera qu’il existe aussi des traductions en allemand, norvégien, suédois, et espagnol ou portugais.
La traduction française du MeSH est utilisée dans la banque de données française LISSA :
Voir aussi l'article : "Les outils documentaires du CHU de Rouen : CISMeF, LiSSa et HeTOP" - BASES n° 378, Février 2020
Le contenu de PubMed hors Medline est constitué de différents types de références :
- des citations non encore indexées avec le MeSH ;
- des citations d’articles hors du domaine biomédical, par exemple la tectonique des plaques ou l’astrophysique, issues de publications prises en compte mais ne couvrant pas seulement le domaine biomédical ;
- des citations d’articles non encore publiés ;
- des citations d’articles parus dans une publication avant qu’elle ne soit indexée dans Medline ;
- des citations antérieures à 1966 qui n’ont pas encore été indexées avec la dernière mise à jour du MeSH ;
- des citations d’articles publiés dans des journaux traitant des sciences de la vie soumis directement à PubMed Central ;
- des citations d’articles de chercheurs présentant les résultats de leurs recherches quand elles ont été financées par le NIH et qui doivent, éventuellement après un certain délai, les mettre à disposition dans un repository ;
- des citations à des livres disponibles dans le NCBI Bookshelf.
Il faut noter que les bases baptisées MEDLINE sur différents serveurs évoqués plus haut ont, en général, un contenu légèrement plus large que le MEDLINE de PubMed puisqu’ils intègrent les citations non encore indexées.
PubMed Central, un complément de PubMed
PubMed Central (PMC), un complément de PubMed, a été créé en 2000.
Il s’agit d’une archive de textes intégraux d’articles publiés dans des revues spécialisées dans le domaine des sciences de la vie.
Certaines de ces revues sont indexées dans MEDLINE, mais ce n’est pas la totalité. On y trouve aussi des articles concernant des recherches menées sur fonds publics, pas nécessairement américains, et qui ont l’obligation, éventuellement après un délai, d’être accessible gratuitement dans un repository en libre accès.
Certains de ces articles sont référencés dans MEDLINE s’ils répondent aux critères de sélection de la NLM.
Pour distinguer les deux, on peut dire que MEDLINE propose une sélection de publications dans le domaine de la littérature biomédicale, tandis que l’objectif de PMC est de proposer une archive pérenne pour des publications concernant des recherches « de bonnes qualité » dans le domaine biomédical.
PMC est d’ailleurs devenue une archive de référence pour différents organismes connus comme la Bill et Melinda Gates Foundation, la Food and Drug Administration (FDA aux USA), ou le Department of Homeland Security.
Cependant le fait que PMC utilise le terme PubMed peut entraîner une confusion, certains pensant que tous les articles présents dans PMC ont été sélectionnés avec la même rigueur que ceux sélectionnés par Medline alors que cela n’est pas le cas. Certains parlent même de PMC comme une backdoor pour PubMed supposée faciliter l’intégration dans PubMed des références de certains articles.
Une partie importante des articles référencés dans PubMed comporte un lien vers PubMed Central et/ou l’éditeur permettant de décharger immédiatement l’intégralité de l’article. Bien souvent, ces articles sont gratuits (en open access) mais cela n’est pas systématiquement le cas.
La nouvelle édition de PubMed
Avant cette dernière mise à jour (voir figure 2.), PubMed en avait déjà connu d’importantes, l’une en 2000 et l’autre en 2010 tandis qu’une version pour mobile est apparue en 2011 mais sur une plateforme séparée avec des possibilités restreintes. En complément à ces mises à jour importantes quelques améliorations ont été installées au fil de l’eau.
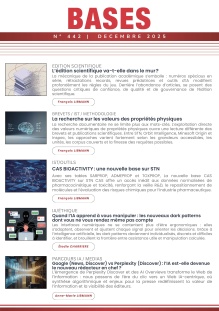
Figure 2. Nouvelle interface de résultats dans Pubmed
Bien que visant un large public dans le monde entier du fait à la fois de sa gratuité (au moins pour les références et en bonne partie pour les articles eux-mêmes) et du domaine couvert qui concerne tout un chacun, PubMed est un produit relativement complexe offrant de multiples possibilités même si on peut aussi l’utiliser de façon basique (un ou deux mots dans la recherche simple suivi de quelques choix ne présentant pas de difficultés).
Les technologies utilisées
Cette nouvelle édition de PubMed a été réalisée en utilisant de nouvelles technologies :
- Solr, un système open source d’indexation et de recherche ;
- MongoDB pour le stockage et la récupération des documents ;
- le Django Web framework pour l’interface utilisateur.
Dans la mesure où ces technologies sont relativement différentes de celles précédemment utilisées et offrent plus de possibilités que celles utilisées dans la précédente plateforme, cela a entraîné des conséquences à la fois sur le nombre de résultats obtenus pour une recherche donnée et sur le classement des résultats.
En particulier, le Automatic Term Mapping qui traduit la recherche initiale en l’enrichissant a été amélioré puisqu’il utilise maintenant aussi les orthographes anglaise et américaine d’un terme, les formes singulier et pluriel ainsi que des synonymes. De plus, la limite de 600 variations pour la troncature a été levée.
Cela se justifie d’autant plus que 80% des utilisateurs ne regardent pas les résultats au-delà de la première page.
Donc, lorsque l’on utilise le nouvel Automatic Term Mapping, on obtient logiquement plus de réponses qu’avec la version précédente et le classement Best Match est modifié en principe dans le sens de l’amélioration de la pertinence. On peut toujours désactiver le Automated Term Mapping en mettant les termes de recherche entre guillemets ou en indiquant un champ, par exemple [tiab] après les termes de recherche. La recherche respectera alors strictement les instructions du chercheur.
Une explication complémentaire pour ces différences de comptage tient au fait que les deux versions de PubMed sont installées sur deux plateformes différentes qui ont des dates différentes d’indexation et aussi d’élimination des doublons et autres opérations.
Notre test
Nous avons fait des tests à quelques minutes d’intervalle dans les deux plateformes en utilisant la recherche
Gene Expression.L’ancienne plateforme donne 1 453 439 réponses tandis que la nouvelle en donne 1 454 852 soit 1 413 documents de plus ce qui représente environ 0,1 % de documents supplémentaires.
Pour les dix premiers résultats classés par pertinence dans la première liste, cinq sont présents à la même place dans la nouvelle version, quatre ont changé de place et un (le dixième) ne figure pas dans la deuxième liste.
Si ce nouvel algorithme permet d’obtenir plus de résultats qui, de plus, sont supposés être plus pertinents, cette rupture est ennuyeuse pour tous ceux qui effectuent une fois par an (fréquence classique) la même recherche pour suivre l’évolution d’un domaine.
On n’a pas cet inconvénient si l’on utilise les serveurs classiques de banques de données tels que STN, Dialog ou Scopus, OVID ou Ebsco. Ceux-ci proposent pour certains des options pour prendre en compte automatiquement les singuliers/pluriels ou les orthographes anglaises/américaines mais ces options sont facilement « débrayables » et ne changent pas, donc il n’y a pas de discontinuité dans la façon de compter les résultats et l’utilisateur garde toujours la main.
Nous avons d’ailleurs découvert des utilisateurs de PubMed, plutôt des professionnels, comme c’est logique, très opposés à la nouvelle plateforme, en particulier pour cette dernière raison (Sciencemag.org, 22 mai 2020 par Michael Price « They redesigned PubMed, a beloved website. It hasn’t gone over well»). Les dirigeants de PubMed répondent, pour leur part, qu’ils sont toujours à l’écoute des feedbacks pour améliorer le produit.
La philosophie, les possibilités et le « look » de cette nouvelle plateforme illustrent l’évolution de PubMed plutôt vers les utilisateurs grand-public ou à tout le moins pas experts plutôt qu’à des professionnels de l’information.
Une nouvelle façon de construire la recherche avancée
Pour en revenir au nouveau PubMed, on notera que la nouvelle recherche avancée permet de construire la recherche en ajoutant étape par étape de nouveaux termes en choisissant à chaque fois l’opérateur souhaité.
Il n’y a toujours pas de possibilité de recherche de proximité, mais si l’on entre plusieurs termes, le système cherche dans son système de subject translation, s’il trouve une phrase correspondante qui peut être un terme du MeSH mais pas nécessairement.
Cela rejoint l’esprit de construction du système de best match dans lequel l’utilisateur n’a pas la pleine maîtrise de sa recherche et est obligé de s’en remettre au système au moins partiellement.
Parmi les nouveautés ou améliorations, on notera sur la partie gauche de la liste de résultats un grand nombre de filtres, sachant que la liste des types de documents peut devenir très détaillée si l’on clique sur additional filters. Il est également beaucoup plus facile d’obtenir la citation de l’article dans différents formats (AMA,MLA,APA,NLM).
On peut aussi découvrir des articles similaires, les articles citants, les liens vers d’autres sources qui sont très variées.
On notera encore la possibilité de classer les résultats par premier auteur ou par publication.
Enfin, signalons que l’interface pour mobile a été significativement améliorée ce qui était considéré comme nécessaire. En fait, la plate-forme relativement peu performante qui était utilisée va être supprimée à l’occasion de la disparition du legacy PubMed et la recherche sur mobile est complètement intégrée à la nouvelle plateforme qui est capable de gérer tous les formats. L’interrogation à partir d’un mobile qui représente aujourd’hui 40% des recherches bénéficie aujourd’hui des mêmes fonctionnalités que celles offertes sur un PC.
Des analogies avec le monde des brevets
Nous n’allons pas détailler ici les multiples possibilités offertes par ce produit, mais son évolution nous rappelle ce que l’on observe depuis de nombreuses années dans le domaine des brevets.
En effet, dans ce domaine, les offices de brevets auprès desquels les demandes de brevets sont déposées ont, parmi leurs missions, l’obligation de faciliter l’accès au public des données qu’ils détiennent comme le NCBI a pour mission de diffuser le plus largement possible l’information biomédicale.
- Dans les deux cas, cela passe par la création et le développement de banques de données en accès gratuit de plus en plus sophistiquées car elles prennent en compte les développements de la technologie.
En parallèle, des sociétés privées s’appuyant sur les mêmes sources ou les mêmes types de sources (pour le biomédical) développent aussi des systèmes de plus en plus sophistiqués mais payants offrant toujours plus de services pour garder une avance sur les services gratuits.
On notera aussi que la NLM ayant pour mission de diffuser les informations biomédicales le plus largement possible, de nombreux systèmes, outre les serveurs cités plus haut, diffusent tout ou partie du contenu de PubMed, gratuitement ou à un prix faible.
- La différence que l’on peut faire avec le monde des brevets est que, dans le cas des brevets, la source est objectivement limitée puisqu’il s’agit des brevets déposés dans l’ensemble des office de brevets dans le monde. Elle ne peut que s’accroitre dans le temps et l’augmentation éventuelle de sources est extrêmement marginale.
En revanche, dans le domaine biomédical, PubMed/Medline n’est qu’une source parmi d’autres, même si elle est importante. En effet, et il existe de nombreuses autres sources dans le domaine telles qu’Embase, Biosis Preview, British Nursing Index et bien d’autres pour ce qui concerne les banques de données. Si certaines des références figurant dans ces bases de données sont aussi présentes dans Medline, c’est loin d’être systématique et un grand nombre n’y figure pas. Il est donc quelque peu restrictif de limiter ses recherches à PubMed/Medline. Mais évidemment les autres banques de données ne sont pas gratuites.
Même s’il est fréquent que certains de leurs contenus soient également dans PubMed, ils offrent de nombreuses autres informations, chères, à quelques exceptions près comme LISSA, déjà citée, qui offre des références à des publications biomédicales écrites en français dont certaines sont dans PubMed mais pas toutes.